Il progetto di sequenziamento del genoma rivela nuovi segreti sull'evoluzione dei gatti
2 Novembre 2023
Questo articolo è stato revisionato secondo il processo editoriale e le politiche di Science X. Gli editori hanno evidenziato i seguenti attributi garantendo la credibilità dei contenuti:
- verifica dei fatti
- pubblicazione con peer review
- fonte affidabile
- correzione di bozze
di Courtney Price, Texas A&M University
I ricercatori della Texas A&M School of Veterinary Medicine & Biomedical Sciences (VMBS) e un team interdisciplinare di collaboratori hanno scoperto nuove informazioni sulla storia dell'evoluzione dei gatti spiegando come i gatti, inclusi i famosi leoni, tigri e gatti domestici, si sono evoluti in diverse specie e gettando luce su come diverse modificazioni genetiche nei gatti siano correlate alle capacità di sopravvivenza come la capacità di annusare la preda.
Confrontando i genomi di diverse specie di gatti, il progetto, pubblicato oggi su Nature Genetics, ha aiutato i ricercatori a capire perché i genomi dei gatti tendono ad avere meno variazioni genetiche complesse (come riarrangiamenti di segmenti di DNA) rispetto ad altri gruppi di mammiferi, come i primati. Ha anche rivelato nuove intuizioni su quali parti del DNA del gatto sono più propense a evolvere rapidamente e come svolgano un ruolo nella differenziazione delle specie.
"Il nostro obiettivo era capire meglio come si sono evoluti i gatti e le basi genetiche delle differenze di carattere tra le specie di gatti", ha detto il Dr. Bill Murphy, professore di integrative bioscienze veterinarie presso la VMBS specializzato nell'evoluzione dei gatti. "Volevamo approfittare di alcune nuove tecnologie che ci permettono di creare mappe genomiche complete dei gatti.
"Le nostre scoperte apriranno porte a coloro che studiano malattie, comportamento e conservazione felina", ha detto. "Lavoreranno con una comprensione più completa delle differenze genetiche che rendono unico ogni tipo di gatto."
Uno dei temi su cui gli scienziati cercavano di ottenere una migliore comprensione è il motivo per cui i cromosomi felini, strutture cellulari contenenti le informazioni genetiche per tratti come il colore del pelo, la taglia e le capacità sensoriali, sono più stabili rispetto ad altri gruppi di mammiferi.
"Da tempo sappiamo che i cromosomi dei gatti tra le specie sono molto simili tra loro", ha detto Murphy. "Ad esempio, i cromosomi dei leoni e dei gatti domestici non differiscono molto affatto. Sembra che ci siano molte meno duplicazioni, riarrangiamenti e altri tipi di variazioni rispetto a quanto comunemente si trova nei grandi scimpanzé".
Nell'ordine dei primati, questo tipo di variazione genetica ha portato all'evoluzione di diverse specie tra cui gli esseri umani e i grandi scimpanzé.
"I genomi dei grandi scimpanzé tendono a rompersi e a riarrangiarsi e persino i genomi umani hanno regioni molto instabili", ha detto Murphy. "Queste variazioni possono predisporre determinati individui a condizioni genetiche come l'autismo e altri disturbi neurologici".
La chiave di questa variazione tra gatti e scimpanzé, come ha scoperto Murphy, sembra essere la frequenza di qualcosa chiamata duplicazioni segmentali - segmenti di DNA che sono copie altamente simili di altri segmenti di DNA presenti altrove nel genoma.
"I ricercatori del genoma dei primati sono stati in grado di collegare queste duplicazioni segmentali ai riarrangiamenti cromosomici", ha detto. "Più duplicazioni segmentali hai nel tuo DNA, più è probabile che i cromosomi si riarrangino, e così via".
"Quello che abbiamo scoperto confrontando un gran numero di genomi di specie di gatti è che i gatti hanno solo una frazione delle duplicazioni segmentali rispetto ad altri gruppi di mammiferi - i primati hanno infatti sette volte più duplicazioni di queste dei gatti. Questa è una grande differenza e ora crediamo di capire perché i genomi dei gatti sono più stabili", ha detto.
Anche se i gatti potrebbero non avere molti grandi riarrangiamenti genetici nel loro DNA, hanno comunque molte differenze. Attraverso la loro ricerca, Murphy e i suoi colleghi comprendono ora meglio quali parti del DNA del gatto causino queste variazioni, in particolare le variazioni che definiscono la speciazione o le differenze tra le specie.
"Risulta che c'è una vasta regione al centro del cromosoma X dove avvengono la maggior parte dei riarrangiamenti genetici", ha detto Murphy. "In effetti, c'è un elemento ripetuto specifico all'interno di questa regione chiamato DXZ4 che le prove ci dicono sia in gran parte responsabile dell'isolamento genetico di almeno due specie di gatti, il gatto domestico e il gatto della giungla".
DXZ4 è ciò che Murphy chiama una ripetizione satellite - non è un gene tipico che codifica per un tratto fisico come il colore del pelo, ma piuttosto, aiuta nella struttura tridimensionale del cromosoma X e probabilmente ha svolto un ruolo importante nella speciazione dei gatti.
'Ancora non conosciamo il meccanismo preciso, ma confrontando tutti questi genomi di gatti, possiamo misurare meglio il tasso di evoluzione di DXZ4 in una specie rispetto alle altre. Quello che abbiamo imparato è che DXZ4 è una delle parti del genoma del gatto che si evolve più rapidamente; si sta evolvendo più velocemente del 99,5% del resto del genoma', ha spiegato.
'A causa del tasso di mutazione, siamo stati in grado di dimostrare perché DXZ4 è probabilmente collegato alla speciazione', ha aggiunto Murphy.
Utilizzando nuovi sequenziamenti di genomi altamente dettagliati, il team ha anche scoperto legami più chiari tra il numero di geni olfattivi, che governano il rilevamento degli odori nei gatti, e la variazione nel comportamento sociale e la relazione con l'ambiente circostante.
'Poiché i gatti sono predatori che si affidano molto all'olfatto per rilevare la preda, il loro senso dell'olfatto è una parte abbastanza importante di ciò che sono', ha detto Murphy. 'I gatti sono una famiglia molto diversa, e abbiamo sempre voluto capire come la variazione genetica giochi un ruolo nell'abilità di diverse specie di gatti di sentire in diversi ambienti.'
'Leoni e tigri hanno una differenza piuttosto grande tra certi geni per odore coinvolti nel rilevamento delle feromoni, che sono sostanze chimiche che gli animali rilasciano nell'ambiente per comunicare informazioni sull'identità, il territorio o il pericolo', ha continuato.
'Pensiamo che la grande differenza sia dovuta al fatto che i leoni sono animali molto sociali che vivono in gruppi familiari, mentre le tigri conducono una vita solitaria. I leoni possono aver ridotto la dipendenza dalle feromoni e da altri odori perché sono costantemente vicini ad altri leoni, come dimostra il minor numero di geni di questo tipo nei loro genomi', ha spiegato.
Le tigri, d'altra parte, devono essere in grado di sentire la preda su territori molto estesi e trovare compagni.
'In generale, le tigri hanno ampi repertori di recettori olfattivi e feromonici', ha spiegato Murphy. 'Pensiamo che questo sia direttamente legato alle dimensioni dei loro territori e alla varietà di ambienti in cui vivono'.
I gatti domestici, d'altra parte, sembra abbiano perso una vasta gamma di geni olfattivi.
'Se non devono viaggiare molto per trovare ciò di cui hanno bisogno perché vivono con le persone, ha senso che la selezione naturale non conservi quei geni', ha detto.
Murphy ha condiviso che il suo esempio preferito nel progetto sono i recettori di odori del gatto pescatore, una specie selvatica di gatto adattata all'ambiente acquatico che vive nel Sud-est asiatico.
'Siamo stati in grado di dimostrare che i gatti pescatori hanno conservato molti geni per rilevare gli odori trasportati dall'acqua, che è una caratteristica piuttosto rara nei vertebrati terrestri', ha detto. 'Tutte le altre specie di gatti hanno perso questi geni specifici nel tempo, ma i gatti pescatori li hanno ancora'.
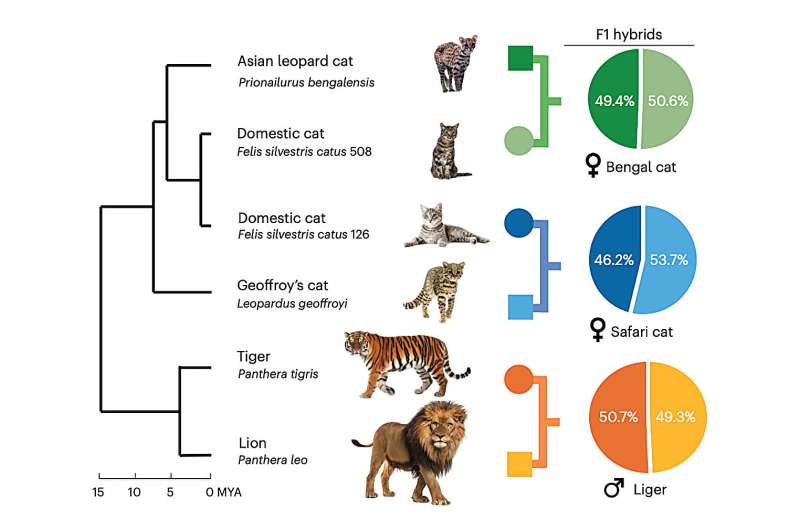
Queste nuove informazioni sui geni olfattivi nei gatti sono state possibili grazie a un nuovo approccio alla sequenziamento del genoma chiamato trio binning, che consente ai ricercatori di sequenziare le regioni più difficili del genoma.
Questa nuova tecnologia rende anche più facile separare il DNA materno e paterno.
'Con il trio binning, ora è possibile prendere il DNA di un ibrido F1 - un animale il cui DNA è diviso al 50% tra i genitori di specie diverse - e separare pulitamente il DNA materno e paterno, ottenendo due set completi di DNA, uno per ciascuna specie genitore', ha detto Murphy. 'Il processo è molto più semplice e i risultati sono più completi'.
Una delle conclusioni più importanti del progetto è che le specie di gatti possono essere simili in molti modi, ma le loro differenze contano.
'Queste differenze ci mostrano come questi animali siano perfettamente adatti ai loro ambienti naturali', ha detto Murphy. 'Non sono interscambiabili, ed è una informazione preziosa per i conservazionisti e per coloro che lavorano per preservare o ripristinare le specie nei loro habitat naturali'.
'Ad esempio, non si può assumere che le tigri di Sumatra e Siberia siano le stesse', ha detto. 'I loro ambienti sono molto diversi, e quelle popolazioni di tigri hanno probabilmente sviluppato adattamenti genetici specializzati per sopravvivere in questi luoghi molto diversi'.
È anche importante per gli scienziati capire che le sezioni dei genomi che sono più difficili da assemblare possono essere la chiave per comprendere sistemi corporei cruciali come l'immunità e la riproduzione.
'I geni olfattivi non sono gli unici che sono stati difficili da sequenziare e studiare. Gli scienziati hanno anche avuto difficoltà a sequenziare geni immunitari e riproduttivi, quindi gli studi precedenti mancano di queste informazioni. Immaginate di cercare di studiare una condizione genetica nei gatti, negli esseri umani o in qualsiasi altra specie, senza avere tutte le informazioni; ecco perché è importante assemblare genomi completi', ha detto Murphy.
Per ora, Murphy e il suo team continueranno ad applicare le più avanzate tecnologie di sequenziamento del genoma e di assemblaggio ai genomi dei gatti per riempire il più possibile le informazioni sul mondo dei gatti.
The study was conceptualized by Bill Murphy—VMBS professor of veterinary integrative biosciences at Texas A&M and Wes Warren—professor of genomics in the Bond Life Sciences Center at the University of Missouri. Additional collaborations involved researchers from the University of Washington, University College Dublin, the Institute for Systems Biology in Seattle, Louisiana State University and the Guy Harvey Oceanographic Center.
Journal information: Nature Genetics
Provided by Texas A&M University




