Das Genomsequenzierungsprojekt enthüllt neue Geheimnisse über die Evolution von Katzen.
2. November 2023
Dieser Artikel wurde gemäß dem Redaktionsprozess und den Richtlinien von Science X überprüft. Die Herausgeber haben die folgenden Attribute hervorgehoben und dabei die Glaubwürdigkeit des Inhalts sichergestellt:
- Faktüberprüfung
- peer-reviewed Veröffentlichung
- vertrauenswürdige Quelle
- Korrektur gelesen
von Courtney Price, Texas A&M University
Forscher an der Texas A&M School of Veterinary Medicine & Biomedical Sciences (VMBS) und ein interdisziplinäres Team von Mitarbeitern haben neue Informationen über die Evolution der Katzen entdeckt und erklärt, wie Katzen - einschließlich bekannter Arten wie Löwen, Tiger und Hauskatzen - sich zu unterschiedlichen Arten entwickelt haben und dabei aufzeigen, wie sich verschiedene genetische Veränderungen bei Katzen auf Überlebensfähigkeiten wie die Fähigkeit zur Geruchswahrnehmung beziehen. Durch den Vergleich der Genome mehrerer Katzenarten hat das Projekt, das heute in Nature Genetics veröffentlicht wurde, den Forschern geholfen zu verstehen, warum Katzen-Genome tendenziell weniger komplexe genetische Variationen (wie Umordnungen von DNA-Segmenten) aufweisen als andere Säugetiergruppen wie Primaten. Es lieferte auch neue Erkenntnisse darüber, welche Teile der Katzen-DNA am wahrscheinlichsten schnell evolvieren und welche Rolle sie bei der Speziesdifferenzierung spielen.
"Unser Ziel war es, besser zu verstehen, wie Katzen sich entwickelt haben und genetische Grundlagen für die Unterschiede zwischen Katzenarten zu finden", sagte Dr. Bill Murphy, Professor für Veterinärmedizinische Integrationsbiowissenschaften an der VMBS und Experte für Katzenentwicklung. "Wir wollten von einigen neuen Technologien profitieren, die es uns ermöglichen, umfassendere genomische Karten von Katzen zu erstellen.
"Unsere Ergebnisse werden Türen für Menschen öffnen, die Katzenkrankheiten, Verhalten und Naturschutz untersuchen", sagte er. "Sie werden mit einem umfassenderen Verständnis der genetischen Unterschiede arbeiten, die jede Katzenart einzigartig machen."
Eines der Dinge, die die Wissenschaftler besser verstehen wollten, ist, warum die Chromosomen der Katzen - zelluläre Strukturen, die genetische Informationen für Merkmale wie Fellfarbe, Größe und sensorische Fähigkeiten enthalten - stabiler sind als in anderen Säugetiergruppen.
"Wir wissen schon seit einiger Zeit, dass Katzenchromosomen zwischen Arten sehr ähnlich sind", sagte Murphy. "Zum Beispiel unterscheiden sich die Chromosomen von Löwen und Hauskatzen kaum voneinander. Es scheint weit weniger Duplikationen, Umordnungen und andere Arten von Variationen zu geben als in den Großen Menschenaffen."
In der Primatenordnung hat diese Art von genetischer Variation zur Evolution von verschiedenen Arten geführt - einschließlich des Menschen und der Großen Menschenaffen.
"Die Genome der Großen Menschenaffen tendieren dazu, zu brechen und sich neu anzuordnen, und sogar die Genome der Menschen haben sehr instabile Regionen", sagte Murphy. "Diese Variationen können dazu führen, dass bestimmte Individuen genetische Erkrankungen wie Autismus und andere neurologische Störungen haben."
Der Schlüssel zu dieser Variation zwischen Katzen und Affen scheint, wie Murphy herausfand, die Häufigkeit von sogenannten segmentalen Duplikationen zu sein - Segmente der DNA, die hochähnliche Kopien anderer DNA-Segmente des Genoms sind.
"Forscher des Primatengenoms konnten diese segmentalen Duplikationen mit Chromosomenumordnungen in Verbindung bringen", sagte er. Je mehr segmentale Duplikationen Sie in Ihrer DNA haben, desto wahrscheinlicher ist es, dass die Chromosomen sich umarrangieren, und so weiter.
"Was wir herausgefunden haben, indem wir eine große Anzahl von Katzenarten-Genomen verglichen haben, ist, dass Katzen nur einen Bruchteil der segmentalen Duplikationen haben, die in anderen Säugetiergruppen zu finden sind - Primaten haben tatsächlich siebenmal mehr dieser Duplikationen als Katzen. Das ist ein großer Unterschied, und jetzt glauben wir, zu verstehen, warum Katzen-Genome stabiler sind", sagte er.
Obwohl Katzen möglicherweise nicht so viele große genetische Umordnungen in ihrer DNA haben, haben sie dennoch viele Unterschiede. Durch ihre Forschung verstehen Murphy und seine Kollegen nun besser, welcher Teil der Katzen-DNA diese Variationen verursacht, insbesondere die Variationen, die die Artenbildung oder die Unterschiede zwischen Arten definieren.
"Es stellt sich heraus, dass es eine große Region auf der Mitte des X-Chromosoms gibt, in der die meisten genetischen Umordnungen stattfinden", sagte Murphy. "In dieser Region gibt es tatsächlich ein spezifisches repetitives Element namens DXZ4, von dem uns die Beweise sagen, dass es hauptsächlich für die genetische Isolation von mindestens zwei Katzenarten, der Hauskatze und der Dschungelkatze, verantwortlich ist."
DXZ4 ist das, was Murphy ein Satellitenrepetitiv nennt - es ist kein typisches Gen, das für ein physisches Merkmal wie Fellfarbe codiert, sondern es trägt zur dreidimensionalen Struktur des X-Chromosoms bei und hat wahrscheinlich eine wichtige Rolle bei der Artenbildung von Katzen gespielt.
Wir kennen noch nicht den genauen Mechanismus, aber indem wir diese Katzen-Genome vergleichen, können wir besser die Geschwindigkeit bestimmen, mit der sich DXZ4 in einer Art im Vergleich zu den anderen entwickelt hat. Was wir gelernt haben, ist, dass DXZ4 einer der am schnellsten entwickelnden Teile des Katzen-Genoms ist; es entwickelt sich schneller als 99,5% des Rests des Genoms", erklärte er.
"Aufgrund der Mutationsrate konnten wir zeigen, warum DXZ4 wahrscheinlich mit der Artbildung verbunden ist", sagte Murphy.
Mit neuen, hochdetaillierten Genomsequenzen entdeckte das Team auch klarere Zusammenhänge zwischen der Anzahl von Riechgengenen, die den Duftdetektion bei Katzen steuern, und der Variation des Sozialverhaltens und wie sie sich auf ihre Umgebung beziehen.
"Da Katzen Raubtiere sind, die stark auf den Geruchssinn angewiesen sind, um ihre Beute aufzuspüren, ist ihr Geruchssinn ein ziemlich wichtiger Teil ihrer Identität", sagte Murphy. "Katzen sind eine sehr vielfältige Familie, und wir wollten schon immer verstehen, wie genetische Variation eine Rolle für die Fähigkeit verschiedener Katzenarten spielt, in ihren unterschiedlichen Umgebungen zu riechen."
"Löwen und Tiger haben einen ziemlich großen Unterschied bei bestimmten Geruchsgenen, die bei der Erkennung von Pheromonen, das sind Chemikalien, die verschiedene Tiere in die Umwelt abgeben, um Informationen über Identität, Territorium oder Gefahr zu kommunizieren, beteiligt sind", fuhr er fort.
"Wir glauben, dass der große Unterschied damit zusammenhängt, dass Löwen sehr soziale Tiere sind, die in Familiengruppen leben, und Tiger ein einzelgängerisches Leben führen. Löwen haben möglicherweise eine geringere Abhängigkeit von Pheromonen und anderen Geruchsstoffen, weil sie ständig in der Nähe von anderen Löwen sind, was sich in der geringeren Anzahl von Genen dieser Art in ihrem Genom widerspiegelt", sagte er.
Tiger hingegen müssen in der Lage sein, Beute in sehr großen Gebieten riechen zu können und auch Partner zu finden.
"Tiger haben im Allgemeinen ein großes Repertoire an Geruchs- und Pheromonrezeptoren", erklärte Murphy. "Wir glauben, dass dies direkt mit der Größe ihrer Territorien und der Vielfalt der Umgebungen, in denen sie leben, zusammenhängt."
Dagegen scheinen Hauskatzen eine breite Palette von Riechgenen verloren zu haben.
"Wenn sie nicht so weit reisen müssen, um das zu finden, was sie brauchen, weil sie mit Menschen leben, ist es sinnvoll, dass die natürliche Selektion diese Gene nicht bewahrt hat", sagte er.
Murphy teilte mit, dass sein Lieblingsbeispiel aus dem Projekt die Geruchsrezeptoren der Fischkatze seien, einer aquatisch angepassten Wildkatzenart, die in Südostasien lebt.
"Wir konnten zeigen, dass Fischkatzen viele Gene zum Nachweis von Geruchsstoffen im Wasser behalten haben, was bei terrestrischen Wirbeltieren eine ziemlich seltene Eigenschaft ist", sagte er. "Alle anderen Katzenarten haben im Laufe der Zeit diese spezifischen Gene verloren, aber Fischkatzen haben sie immer noch."
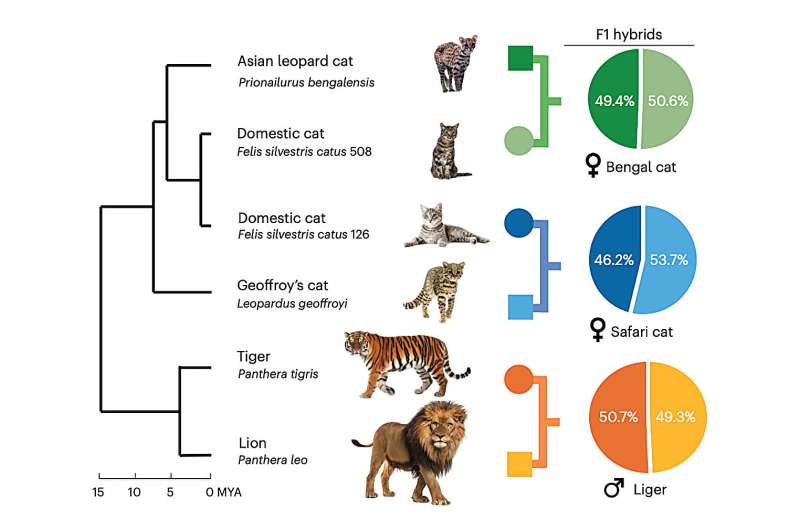
Diese neuen Informationen über Riechgengene bei Katzen wurden durch einen neuen Ansatz zur Genomsequenzierung namens Trio Binning möglich, der es Forschern ermöglicht, die schwierigsten Regionen eines Genoms zu sequenzieren.
Diese neue Technologie erleichtert auch die Trennung von mütterlicher und väterlicher DNA.
"Mit dem Trio Binning können Sie jetzt DNA von einem F1-Hybriden nehmen - einem Tier, dessen DNA zu gleichen Teilen von Eltern unterschiedlicher Arten stammt - und die mütterliche und väterliche DNA sauber trennen, so dass Sie zwei vollständige DNA-Sätze haben, einen für jede Elternart", sagte Murphy. "Der Prozess ist viel einfacher und die Ergebnisse sind umfassender."
Eine der wichtigsten Schlussfolgerungen aus dem Projekt ist, dass Katzenarten in vielerlei Hinsicht ähnlich sein können, aber ihre Unterschiede wichtig sind.
"Diese Unterschiede zeigen uns, wie perfekt diese Tiere für ihre natürlichen Lebensräume geeignet sind", sagte Murphy. "Sie sind nicht austauschbar, und das ist wertvolle Information für Naturschützer und andere, die daran arbeiten, Arten in ihren natürlichen Lebensräumen zu erhalten oder wiederherzustellen.
"Zum Beispiel darf man nicht davon ausgehen, dass Tiger aus Sumatra und Sibirien gleich sind", sagte er. "Ihre Lebensräume sind stark unterschiedlich, und diese Tigerpopulationen haben wahrscheinlich spezialisierte genetische Anpassungen entwickelt, um in diesen sehr unterschiedlichen Orten zu überleben."
Es ist auch wichtig für Wissenschaftler zu erkennen, dass die Abschnitte von Genomen, die am schwersten zusammenzusetzen sind, möglicherweise der Schlüssel zum Verständnis wichtiger Körpersysteme wie Immunität und Fortpflanzung sind.
"Geruchsgene sind nicht die einzigen, die schwierig zu sequenzieren und zu untersuchen waren. Wissenschaftler haben auch Schwierigkeiten, Immun- und Reproduktionsgene zu sequenzieren, daher fehlen in früheren Studien diese Art von Informationen. Stellen Sie sich vor, Sie versuchen, eine genetische Erkrankung bei Katzen, Menschen oder einer anderen Art zu untersuchen, ohne alle Teile zu haben; deshalb sind vollständige Genome von Bedeutung", sagte Murphy.
Vorerst werden Murphy und sein Team weiterhin die fortschrittlichsten Genomsequenzierungs- und Assembliertechnologien auf Katzen-Genomen anwenden, um möglichst viele Informationen über die Welt der Katzen zu erhalten.
The study was conceptualized by Bill Murphy—VMBS professor of veterinary integrative biosciences at Texas A&M and Wes Warren—professor of genomics in the Bond Life Sciences Center at the University of Missouri. Additional collaborations involved researchers from the University of Washington, University College Dublin, the Institute for Systems Biology in Seattle, Louisiana State University and the Guy Harvey Oceanographic Center.
Journal information: Nature Genetics
Provided by Texas A&M University




