Un kit per l'assemblaggio del DNA per sbloccare il potenziale di CRISPR-Cas9 nell'ingegneria metabolica.
30 agosto 2023 feature
Questo articolo è stato revisionato secondo il processo editoriale e le politiche di Science X. Gli editor hanno evidenziato i seguenti attributi garantendo la credibilità del contenuto:
- fatto il controllo dei fatti
- pubblicazione sottoposta a revisione paritaria
- fonte affidabile
- corretto
di Thamarasee Jeewandara, Phys.org
La tecnica dei ripetuti palindromi corti intervallati in cluster (CRISPR) e la proteina associata Crispr 9 (CRISPR/Cas9) sono ora metodi rivoluzionari ben noti per ingegnerizzare cellule microbiche.
Un vantaggio chiave di CRISPR rimane nella progettazione del ceppo per facilitare l'integrazione cromosomica per consentire l'assemblaggio di DNA privo di marcatori. Questi sistemi di modifica sono molto vantaggiosi; tuttavia, la loro assemblaggio non è così semplice e potrebbe impedire il suo utilizzo e le sue applicazioni.
In un nuovo rapporto su Nature Communications Biology, Tigran V. Yuzbashev e un team di ricerca hanno identificato i limiti degli attuali strumenti Cas9 progettati per rendere più facile l'accesso e l'implementazione delle tecniche CRISPR. Hanno discusso tre diversi metodi ben stabiliti e li hanno combinati per formare un toolkit completo per l'ingegneria metabolica efficiente mediante l'uso di CRISPR/Cas9.
Un unico toolkit composto da 147 plasmidi per generare e caratterizzare una libreria di 137 promotori per costruire acido omogenitico in laboratorio.
Il sistema CRISPR/Cas9 può apportare modifiche genomiche rapide, precise e senza cicatrici per fornire un ampio spettro di progettazione di ceppi microbici per la bioproduzione. L'ingegneria metabolica dei lieviti, ad esempio, rappresenta un'area di crescita rapida nell'ingegneria biologica per la produzione sostenuta di prodotti chimici, carburanti, alimenti e farmaci.
I lieviti hanno un potenziale metabolico simile alle cellule eucariotiche e sono quindi più facili da ingegnerizzare e coltivare su larga scala. Di conseguenza, gli bioingegneri hanno progettato e sviluppato sistemi CRISPR per i lieviti.
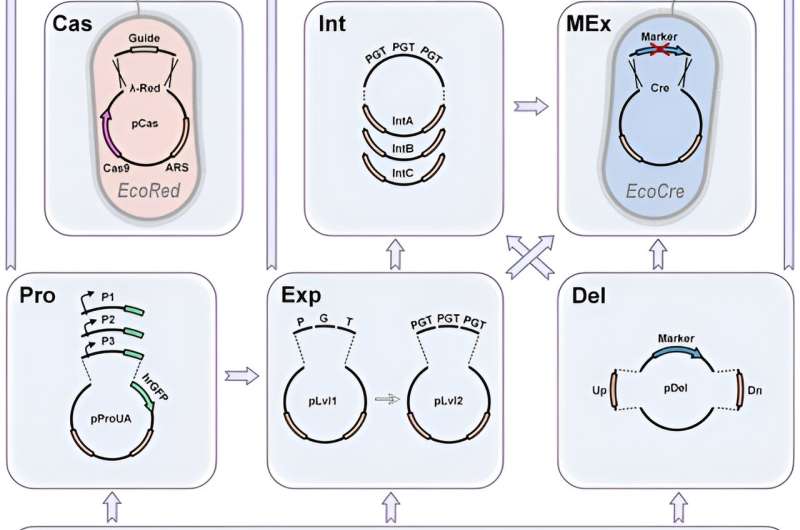
Grazie alla sua elevata efficienza, CRISPR consente modifiche genomiche senza marcatori. In questo lavoro, Yuzbashev ha garantito l'ottimizzazione del ceppo e ha facilitato i progetti di ingegneria metabolica identificando tre miglioramenti del sistema CRISPR/Cas9 per l'ingegneria dei lieviti. I metodi includono: 1) il cambio semplice tra modifiche con marcatori e senza marcatori, 2) lo scambio rapido di bracci di omologia per determinare diverse posizioni di integrazione, e 3) un metodo semplice per clonare le gRNA.
Per consentire l'integrazione basata su CRISPR senza marcatori, il team ha scelto una rottura del doppio filamento indotta da Cas9, che doveva essere riparata per permettere la proliferazione delle cellule. I ricercatori sono riusciti a realizzare ciò utilizzando un modello o donatore, integrato attraverso ricombinazione omologa o unione di estremità non omooghe (NHEJ), senza integrazione. Il processo di unione di estremità non omologhe è osservato nella maggior parte delle specie fungine, compreso il lievito del pane Saccharomyces cerevisiae.
In specie con un meccanismo NHEJ predominante, il team ha potenziato la ricombinazione omologa eliminando i geni NHEJ. Se un metodo senza marcatori non avesse avuto successo, i ricercatori avrebbero successivamente cercato di migliorare l'integrazione assistita da CRISPR-Cas9 per tornare facilmente all'integrazione basata su marcatori.
L'integrazione assistita da Cas9 richiede tipicamente un modello di donatore composto da una cassetta integrata flanccata da due bracci di omologia. Il team ha ipotizzato che l'integrazione ideale di CRISPR/Cas9 dovesse scambiare bracci di omologia su costrutti donatori Cas9 pre-valutati tramite una semplice reazione di assemblaggio Golden Gate.
Inoltre, i promotori sono un elemento chiave per qualsiasi progetto di ingegneria metabolica per indirizzare il flusso verso i prodotti di interesse. Yuzbashev et al. hanno utilizzato il lievito industriale Yarrowia lipolytica per sviluppare un toolkit di ingegneria metabolica che combinava modifiche genetiche e strategie di assemblaggio del DNA per elevate efficienza e versatilità.
I ricercatori hanno sfruttato appieno il potenziale di CRISPR/Cas9 per l'ingegneria metabolica sviluppando un toolkit che ampliava i sistemi di assemblaggio Golden Gate precedentemente noti. Hanno testato il sistema di screening generando diverse librerie di promotori. Yuzbashev et al. hanno scelto i geni ribosomiali di Y. lipolytica che codificano proteine di subunità grandi e piccole. Hanno identificato una varietà di promotori con diverse intensità per ampliare il numero di promotori per lo stesso organismo.
Per dimostrare l'influenza e l'utilizzo del metodo migliorato CRISPR/Cas9, il team ha creato un Y. lipolytica tramite ingegneria razionale per produrre un acido omogenitico (HGA). Tipicamente in condizioni alcaline, l'HGA subisce spontaneamente ossidazione per formare piro-melanina autopolimerizzata; un ottimo componente di creme solari naturali e cosmetici.
Despite its high commercial potential, existing methods to produce the acid precursor and pyomelanin product relied on the biotransformation of expensive aromatic amino acids. To facilitate metabolic engineering, the team therefore first selected several genes that encoded theprecursor aromatic aminotransferases as engineering targets. They then selected three overexpression targets to enhance the de novo synthesis of the homogentisic acid in the model organism. Finally, they studied and inactivated the HGA degradation pathway; a path yet unknown to exist in Y. lipolytica.
In this way, Tigran V. Yuzbashev and colleagues showed the dependence of metabolic engineering of living organisms on efficient DNA manipulation methods. This work presents an example of an enhanced molecular toolkit designed for CRISPR/Cas9-based metabolic engineering.
The scientists proved the functionality of the platform for both rapid strain construction and the characterization of a large library of promoters. They anticipate for this toolkit to have broader applications in strain engineering and in industry. The team envision for the Y. lipolytica model developed in this work to have overarching applications in other fields of biological engineering as well.
Guri Giaever et al, Functional profiling of the Saccharomyces cerevisiae genome, Nature (2002). DOI: 10.1038/nature00935
Journal information: Nature , Communications Biology
© 2023 Science X Network




