Un kit d'assemblage d'ADN pour exploiter le potentiel de CRISPR-Cas9 pour l'ingénierie métabolique.
30 août 2023
caractéristique
Cet article a été examiné selon le processus éditorial et les politiques de Science X. Les éditeurs ont mis en évidence les attributs suivants tout en veillant à la crédibilité du contenu :
- vérification des faits
- publication évaluée par les pairs
- source fiable
- relecture
par Thamarasee Jeewandara, Phys.org
Les répétitions régulièrement groupées de palindromes courts espacés (CRISPR) et la protéine associée à CRISPR 9 (CRISPR/Cas9) sont maintenant une méthode révolutionnaire bien connue pour modifier les cellules microbiennes.
Un avantage clé de CRISPR réside dans la conception de la souche pour faciliter l'intégration chromosomique afin de permettre l'assemblage d'ADN sans marqueur. Ces systèmes de modification sont extrêmement bénéfiques ; cependant, leur assemblage n'est pas aussi simple et peut empêcher leur utilisation et leurs applications.
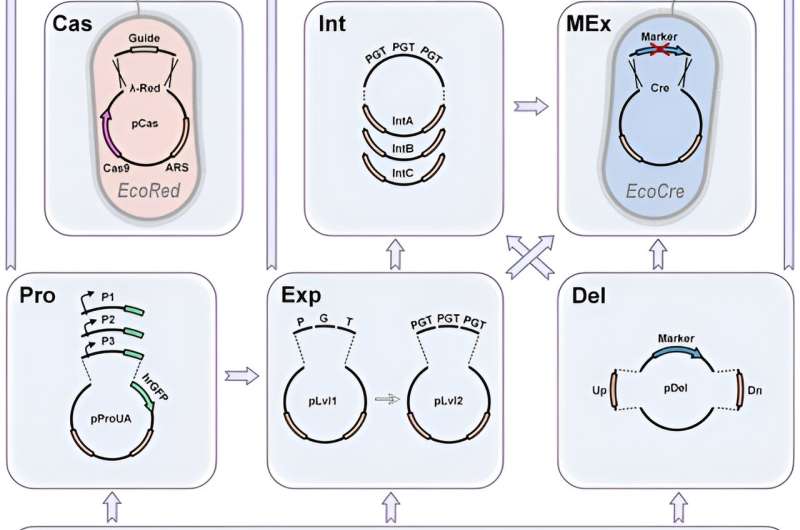
Dans un nouveau rapport publié dans Nature Communications Biology, Tigran V. Yuzbashev et une équipe de recherche ont identifié les limites des boîtes à outils Cas9 existantes conçues pour faciliter l'accès et la mise en œuvre des techniques CRISPR. Ils ont discuté de trois méthodes différentes bien établies et les ont combinées pour former une boîte à outils complète pour l'ingénierie métabolique efficace en utilisant CRISPR/Cas9.
Une seule boîte à outils comprenait 147 plasmides pour générer et caractériser une bibliothèque de 137 promoteurs permettant de construire de l'acide homogentisique en laboratoire.
Le système CRISPR/Cas9 peut effectuer des modifications génomiques rapides, précises et sans cicatrice pour offrir une grande possibilité de concevoir des souches microbiennes pour la bioproduction. L'ingénierie métabolique des levures, par exemple, constitue un domaine en plein essor de l'ingénierie biologique pour la production durable de produits chimiques, de carburants, d'aliments et de produits pharmaceutiques.
Les levures ont un potentiel métabolique similaire à celui des cellules eucaryotes et sont donc plus faciles à manipuler et à cultiver à grande échelle. En conséquence, les bioingénieurs ont conçu et développé des systèmes CRISPR pour les levures.
Grâce à son efficacité élevée, CRISPR permet des modifications génomiques sans marqueur. Dans ce travail, Yuzbashev a assuré l'optimisation de la souche et facilité les projets d'ingénierie métabolique en identifiant trois améliorations du système CRISPR/Cas9 pour l'ingénierie des levures. Les méthodes comprenaient : 1) le remplacement facile entre les modifications avec et sans marqueur, 2) l'échange rapide de bras d'homologie pour déterminer différents emplacements d'intégration, et 3) une méthode facile pour cloner les gARN.
Pour permettre l'intégration sans marqueur basée sur CRISPR, l'équipe a choisi une cassure double brin induite par Cas9, qui devait être réparée pour assurer la prolifération cellulaire. Les scientifiques ont rendu cela possible en utilisant un modèle ou un donneur, intégré par recombinaison homologue ou jonction d'extrémités non homologues (NEHJ) - sans intégration. Le processus de jonction d'extrémités non homologues est observé dans la plupart des espèces fongiques, y compris la levure de boulanger Saccharomyces cerevisiae.
Dans les espèces avec un mécanisme NHEJ prédominant, l'équipe a amélioré la recombinaison homologue en supprimant les gènes NHEJ. Si une méthode sans marqueur n'a pas réussi, les scientifiques visent ensuite à améliorer l'intégration assistée par CRISPR-Cas9 pour revenir facilement à une intégration basée sur marqueur.
L'intégration assistée par Cas9 nécessite généralement un modèle donneur composé d'une cassette intégrée flanquée de deux bras d'homologie. L'équipe a postulé que l'intégration idéale de CRISPR/Cas9 devrait échanger les bras d'homologie sur des constructions donneuses Cas9 pré-évaluées via une réaction d'assemblage Golden Gate simplifiée.
De plus, les promoteurs sont un élément clé de tout projet d'ingénierie métabolique pour rediriger le flux vers les produits d'intérêt. Yuzbashev et al. ont utilisé la levure industrielle Yarrowia lipolytica pour développer une boîte à outils d'ingénierie métabolique combinant l'édition de gènes et des stratégies d'assemblage d'ADN pour une efficacité et une polyvalence élevées.
Les scientifiques ont exploité le plein potentiel de CRISPR/Cas9 pour l'ingénierie métabolique en développant une boîte à outils qui s'appuie sur des systèmes d'assemblage Golden Gate déjà bien connus. Ils ont testé le système de screening en générant plusieurs bibliothèques de promoteurs. Yuzbashev et al. ont choisi les gènes de ribosomes de Y. lipolytica codant pour des protéines de grandes et petites sous-unités. Ils ont identifié une variété de promoteurs avec des forces différentes pour augmenter le nombre de promoteurs pour le même organisme.
Pour prouver l'influence et l'utilisation de la méthode améliorée CRISPR/Cas9, l'équipe a créé une Y. lipolytica par génie rationnel pour produire de l'acide homogentisique (HGA). En conditions alcalines, l'HGA subit spontanément une oxydation pour former de la pyomélanine autopolymérisée ; un excellent constituant des écrans solaires naturels et des cosmétiques.
Despite its high commercial potential, existing methods to produce the acid precursor and pyomelanin product relied on the biotransformation of expensive aromatic amino acids. To facilitate metabolic engineering, the team therefore first selected several genes that encoded theprecursor aromatic aminotransferases as engineering targets. They then selected three overexpression targets to enhance the de novo synthesis of the homogentisic acid in the model organism. Finally, they studied and inactivated the HGA degradation pathway; a path yet unknown to exist in Y. lipolytica.
In this way, Tigran V. Yuzbashev and colleagues showed the dependence of metabolic engineering of living organisms on efficient DNA manipulation methods. This work presents an example of an enhanced molecular toolkit designed for CRISPR/Cas9-based metabolic engineering.
The scientists proved the functionality of the platform for both rapid strain construction and the characterization of a large library of promoters. They anticipate for this toolkit to have broader applications in strain engineering and in industry. The team envision for the Y. lipolytica model developed in this work to have overarching applications in other fields of biological engineering as well.
Guri Giaever et al, Functional profiling of the Saccharomyces cerevisiae genome, Nature (2002). DOI: 10.1038/nature00935
Journal information: Nature , Communications Biology
© 2023 Science X Network




